Augmented Chemistry

Scientific Director: GudrunKlinker
Contact Person(s): Patrick Maier
| Start: | 1 October 2007 |
| End: | 1 January 2010 |
Keywords
Keywords: Augmented Chemistry molecules visualisation bindings XNA TINKER UbiTrack
Abstract
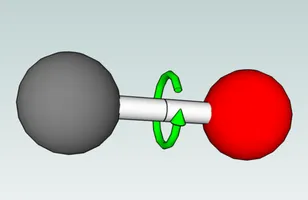
The aim of this project is to help chemists to create, visualize molecules and chemical reactions. It will be possible to see if molecules have enough space to react with eachother. The molecules are rotated and placed with markers in the real world and displayed on a monitor or on vr-glasses.
Pictures
Videos
Video 01:
http://campar.in.tum.de/files/movies/flash_movies/ProjectChemistry.flv
Video 02:
http://campar.in.tum.de/files/movies/flash_movies/ProjectChemistry2.flv
Publications
Team
Contact Person(s)

Interaktive Chemie Visualisierung DEMO
Demo starten
- chemvis.zip: Demoprogramm fuer Augmented Chemistry downloaden
- In ein neues Verzeichnis entpacken
- Marker-Cube.pdf ausdrucken und den Würfel zusammenkleben
- Webcam anschließen und OSG_Test.exe ausführen
- Folgende Marker in das Kammerabild halten.
Marker für die Moleküle - Viel Spaß :-)
Programm
- Struktur des Szenengraphen
- Das Molekül besteht aus einzelnen Instanzen von Atom-Klassen
- Die Atom-Klassen bestehen aus einer Kugel (Atom) und den Bindungen
- Wenn ein Atom noch keine Bindung eingegangen ist, dann stehen alle Bindungen in eine Richung aus dem Atom heraus (in richtung der x-Achse des Atom-Koordinatensystems)
- Diese Bindungen haben eine bestimmte Länge (kurzer Stummel) in einer bestimmten Farbe (erst einmal grün)
- Wenn 2 Atomklassen miteinander verbunden sind, dann berühren sich die 2 Bindungen in der Mitte und ergeben eine bestimmte Länge, die durch die Kombination der unterschiedlichen Atomklassen vorgegeben sind.
- Um dieses Berührungspunkt können sich die beiden Atom-Klassen zueinander verdrehen.
- Wenn eine zusätzliche Atom-Klasse zu der ersten kommt, dann zeigen ab einem bestimmten Abstand die restlichen Bindungen zu dieser atom-Klasse hin.
- Wenn alle Bindungen der ersten Atom-Klasse gebunden sind, dann ordnen sich die einzelnen Bindungen in bestimmten Winkeln zueinader an, die in Abhängigkeit der gekoppelten Atom-Klassen steht. (Die Winkel werden aus einer Datenbank genommen)
Location

Technische Universität München
Institut für Informatik / I16
Boltzmannstr. 3
85748 Garching bei München
Tel.: +49 89 289-17058
Fax: +49 89 289-17059